Charakterystyka proteinazy K, aktywność enzymatyczna i zastosowania
The proteinaza K jest enzymem należącym do grupy proteaz serynowych, to znaczy ma w swoim aktywnym centrum katalitycznym aminokwas serynowy i ma funkcję rozbijania wiązań peptydowych przez hydrolizę. Z kolei ten enzym należy do rodziny białek subtylizyn (peptydaza S8).
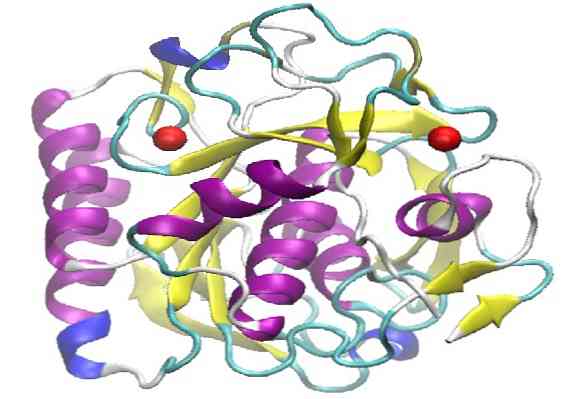
Proteinaza K ma masę cząsteczkową (MW) 28 900 daltonów i została wyizolowana po raz pierwszy w 1974 r. Z ekstraktów grzyba Album Engyodontium, wcześniej znany pod nazwą Tritirachium album Limber.
Wykazuje wysoką zdolność proteolityczną, wykazując zdolność do degradacji keratyny obecnej we włosach. Słowo keratyna w języku angielskim jest napisane „keratyna”, dlatego zostało nazwane „proteinazą K”.
Ze względu na wysoką zdolność rozszczepiania natywnych białek enzym ten jest przydatny w różnych technikach biologii molekularnej. Służy głównie do izolacji i przygotowania kwasów nukleinowych o wysokiej masie cząsteczkowej (MW).
Proteinaza K działa poprzez uwalnianie jądrowego DNA, niszcząc białka i dezaktywuje RNazy i DNazy, to znaczy eliminuje nukleazy w preparatach DNA i RNA..
Z drugiej strony zaobserwowano, że proteinaza K może hydrolizować niektóre zdenaturowane białka natywne, co wzbudziło zainteresowanie naukowców do wykorzystania w badaniu białek prionowych (PrPC).
Jednak pomimo wysokiej siły proteolitycznej istnieją białka, które są odporne na działanie proteinazy K. Wśród nich są pewne nieprawidłowe białka zwane prionami (PrPSc), związane z pasażowalnymi encefalopatiami gąbczastymi.
Indeks
- 1 Charakterystyka proteinazy K
- 2 Aktywność enzymatyczna
- 3 aplikacje
- 4 Zalety proteinazy K
- 5 Białka odporne na proteinazę K
- 6 referencji
Charakterystyka proteinazy K
Proteinaza K ma trzeciorzędową strukturę utworzoną przez trzy warstwy, z arkuszem β siedmiu łańcuchów przeplatanym między dwiema warstwami helis. Ponieważ należy do rodziny peptydaz S8, charakteryzuje się triadą katalityczną w miejscu aktywnym, której kolejność jest następująca (Asp, His i Ser), co odróżnia je od innych rodzin peptydaz..
Ten enzym z grupy proteaz serynowych charakteryzuje się hydrolizą wiązań peptydowych blisko grupy karboksylowej aminokwasów alifatycznych i aromatycznych.
Z drugiej strony, jest on zdolny do działania w obecności pewnych żrących substancji, takich jak dodecylosiarczan sodu (SDS), Tris-HCL i EDTA, które są używane do pomocy w denaturacji białek, powodując ich utratę naturalnej struktury.
Jest to wstępny krok w przygotowaniu białek do techniki elektroforezy. Zakres pH, w którym działa proteinaza K, jest dość szeroki (2,0 do 12,0), z optymalnym pH między 7,5 a 12,0, a jego punkt izoelektryczny wynosi 8,9. Jak można zaobserwować, jest on aktywny w bardzo szerokim zakresie pH.
Inną cechą wyróżniającą proteinazę K jest jej stabilność w obecności wysokich temperatur (50–60 ° C).
Aktywność enzymatyczna
Proteinaza K potrzebuje obecności jonów wapnia, chociaż nie wpływa to na jej aktywność, jeśli jest to niezbędne do utrzymania jej stabilności.
Aby proteinaza K mogła przeprowadzić całkowite trawienie substratu, potrzebny jest przybliżony czas kontaktu od 5 minut do 2 godzin.
Jednak w tym sensie Daza i wsp. Porównali czystość DNA uzyskanego przy kilkukrotnej ekspozycji na proteinazę K i doszli do wniosku, że przedłużona inkubacja (do 24 godzin) znacząco poprawia jakość DNA.
Teraz, w odniesieniu do stężenia używanego enzymu proteinazy K w różnych protokołach, można powiedzieć, że jest on bardzo zróżnicowany.
Można go stosować od bardzo niskich stężeń (5 μg / ml) do stężeń 500 μg / ml. Jednak najczęściej występujące stężenia robocze wahają się od 50-100 μg / ml, szczególnie w przypadku trawienia białek i inaktywacji nukleaz. Chociaż do leczenia tkanek wymagane jest stężenie 2 mg / ml.
Aplikacje
Jego zastosowania są bardzo szerokie i można je podsumować w następujący sposób:
-Jest on stosowany w trawieniu białek i ekstrakcji DNA za pomocą kilku metod, takich jak: wysalanie, PK-SDS, bromek cetylotrimetyloamoniowy (CTAB), modyfikowany octan potasu i ekstrakcja jodkiem sodu..
-Inaktywacja nukleaz (RNazy i DNazy).
-W technice hybrydyzacji in situ (HIS), aby pomóc uwolnić kwas nukleinowy, oprócz eliminacji niepożądanych białek.
-Modyfikacja białka.
-Na poziomie badawczym, w różnych badaniach.
Zalety proteinazy K
Przeprowadzono kilka badań porównawczych wśród technik ekstrakcji DNA przy użyciu proteinazy K, a inne, które ich nie stosują, wyciągają wnioski, że przy stosowaniu enzymu istnieją większe korzyści. Wśród zalet można wymienić:
-Uzyskuje się DNA o wysokiej masie cząsteczkowej o wysokiej jakości i czystości.
-Wyekstrahowany DNA jest stabilny do 3 miesięcy.
Wyekstrahowane DNA można stosować w następujących technikach: Southern blot, reakcja łańcuchowa polimerazy (PCR), między innymi elektroforeza.
Białka odporne na proteinazę K
Różne badania wykazały, że priony (nieprawidłowe białka toksyczne PrPSc) różnią się od białek PrPC (natywne), ponieważ są odporne na działanie proteinazy K, podczas gdy PrPC są wrażliwe na ich działanie.
Inni autorzy opisali, że w strukturze PrPSc są wrażliwe części i inne odporne na proteinazę K. Jednak obie części są równie toksyczne i zakaźne..
Z drugiej strony Bastian i współpracownicy w 1987 r. Wyizolowali 4 białka 28, 30, 66 i 76 kda z gatunku Spiroplasma mirum. Wszystkie były odporne na działanie proteinazy K i miały także reakcję krzyżową z niektórymi prionami.
Wiadomo, że gatunek ten może powodować zaćmę i istotne uszkodzenia neurologiczne, a ze względu na naukowe odkrycia Bastiana, wśród innych badań, podjęto próbę powiązania tego mikroorganizmu z pasażowalnymi encefalopatiami gąbczastymi.
Jednak etiologia tej zwyrodnieniowej patologii neurologicznej jest nadal przypisywana prionom dzisiaj.
W tym sensie Butler i współpracownicy w 1991 r. Zidentyfikowali i scharakteryzowali klasę białka K 40 odpornego na proteinazę K z dwóch szczepów Mycoplasma hyorhinis. Ten patogen atakuje świnie, infekując ich tkanki, ale w tym przypadku nie było reakcji krzyżowej z badanymi prionami.
Potrzeba więcej badań, aby wyjaśnić wiele niewiadomych na ten temat.
Referencje
- Bastian F, Jennings R i Gardner W. 1987. Antysurowica na białko włókniste związane ze scrapie reaguje krzyżowo z Spiroplasma mirum białka włókniste. J. Clin. Microbiol. 25: 2430-2431.
- Daza C, Guillen J, King J, Ruiz V. Ocena metody ekstrakcji i oczyszczania DNA z tkanki mięśniowej utrwalonej w formaldehydzie z niezidentyfikowanych zwłok. Med Magazine, 2014; 22 (1): 42-49,
- Butler G, Kotani H, Kong L, Frick M, Evancho S, Stanbridge E i Mcgarrity G. Identyfikacja i charakterystyka białek opornych na proteazę K u członków klasy Mollicutes. Infection and Immunity, 1991, 59 (3): 1037-1042
- López M., Rivera M., Viettri M., Lares M., Morocoima A, Herrera L, et al. Porównanie dwóch protokołów ekstrakcji DNA z Trypanosoma cruzi wyhodowane w ośrodku aksenicznym. Rev. Peru. Med. Exp. Public Health 2014; 31 (2): 222-227. Dostępne pod adresem: scielo.org
- Jiménez G, Villalobos M, Jiménez E i Palma W. Określenie skuteczności pięciu protokołów ekstrakcji DNA z materiału parafinowego do badań molekularnych. Rev Méd Univ Costa Rica. 2007; 1 (1): 10-19.


